install.packages("rgbif")
library(rgbif)Na teh vajah boste:
- spoznali bazo podatkov Global Biodiversity Information Facility (GBIF),
- se naučili instalirati in uporabiti dodatne R funkcije (iz paketov
rgbif,dplyrinleaflet), - uporabili R ukaze za dostop do in prenos lokacij iz GBIF serverja ,
- se naučili izbiranja stolpcev in filtiranja podatkov iz tabel,
- se naučili prikazati lokacije točk z uporabo paketa
leafletin izvoziti interaktivni zemljevid vhtmlformat.
Kaj je Global Biodiversity Information Facility (GBIF)?
Global Biodiversity Information Facility (GBIF) je mednarodno omrežje oragnizacija, ki je vzpostavilo podatkovno infrastrukturo oziroma bazo. Financirajo ga vlade držav, namenjen pa je prostemu dostopu do podatkov o pojavljanju vrst na našem planetu.
Ob času pripravljanja teh vsebin je bilo na portalu objavljenih več kot 2.2 milijardi lokacij, tako v morskih kot tudi kopenskih ekosistemih na globalni ravni. Že sama spletna stran je zelo uporabna za iskanje in prenos podatkov, še boljše pa je, da lahko podatke o vrstah ali taksonih poiščemo in prenesemo z uporabo R ukazov neposredno iz serverja GBIF-ja.
Odprite spletno stran GBIF in jo raziščite v naslednjih nekaj minutah.
Dostop do podatkov v spletni bazi GBIF v R-u
Podatke iz GBIF je mogoče prenesti ročno in nato uvoziti v R. Vendar če so programerji lahko leni, smo lahko leni tudi biologi, kajne? V ta namen obstaja paket R, imenovan rgbif. Vsebuje funkcije, ki nam omogočajo neposreden stik s strežnikom GBIF, komuniciranje z njim in pridobivanje podatkov, ki nas zanimajo. V R-U moramo najprej inštalirati oziroma namestiti paket s funkcijo install.packages() z imenom paketa v narekovajih znotraj oklepajev. To samodejno prenese in namesti paket. Prav tako moramo paket naložiti vsakič, ko zaženemo ali znova zaženemo R ali RStudio. To naredimo s funkcijo library() z imenom paketa brez narekovajev v oklepaju. Vsakemu paketu je priložen dokument, v katerem so na voljo vse pomembne informacije, primer za rgbif je dostopen tukaj.
Z instaliranjem osnovnega R-a so nam na voljo le nekatera privzeta orodja. Razlog za to je, da obstaja tako veliko število orodij, da jih posamezen uporabnik velike večine nikoli ne bo potreboval. Lahko pa enostavno dodajamo tista, ki jih potrebumo, tako da dodatne “pakete” prenesemo iz interneta in inštaliramo na naš računalnik (predstavljajte si R paket kot razširitev osnovnega R-a). Inštalacijo paketa potrebujemo narediti le enkrat, pred prvo uporabo le-tega!
Pozorni pa moramo biti, da pred vsako uporabo paketa, le-tega naredimo dostopnega za uporabo, kar pomeni, da ga moramo aktivirati vsakič, ko odpremo R ali RStudio!
Odprite nov R skript v meniju File tako, da kliknete New file in potem R script. Skript bo imel ime Untitled1 (ali Untitled2, če je že odprt prazen skript).
Shranite skript na namizje vašega računalnika in ga poimenujte Dostop do podatkov iz portala GBIF z R: braszdasti kit.
Kopirajte in prilepite spodnjo kodo v skript R, premaknite kazalec v prvo vrstico s kodo in kliknite Run. Premaknite kazalec na vsako vrstico, ki vsebuje kodo, in ponovite.
Če vam R javi napako:
Error in library(...) : there is no package called ‘...’: paket ni inštaliran na računalniku in morate zagnati funkcijoinstall.packages("..."), kjer...predstavljajo ime želenega paketa.Error in ... : could not find function "...": paket ni aktiviran in morate zagnati funkcijolibrary("..."), kjer...predstavljajo ime želenega paketa.
Za dostop do podatkov o pojavljanju vrst obstaja funkcija occ_search(), ki bo vzpostavila stik s strežnikom GBIF, poiskala in prenesla podatke iz baze podatkov. Za primer bomo vzeli brazdastega kita (Balaenoptera physalus).

Funkcije ste spoznali na prvih vajah, kjer smo znotraj oklepajev natipkali ime nekega objekta, da smo dobili rezultat. Poleg objektov, ki jih ustvarimo, funkcije sprejemajo tudi druge vnose, ki jih imenujemo argumenti. Funkcija occ_search() ima med mnogimi možnimi argumenti, enega poimenovanega scientificName =, ki je privzeto nastavljen na NULL. Če želimo uporabiti funkcijo occ_search() za iskanje pojavljanj posamezne vrste, kot je naš primet brazdastega kita, argument izenačimo z imenom iskane vrste: scientificName = "Balaenoptera physalus". Upoštevajte, da je latinsko ime napisano v narekovajih, da ga R obravnava kot besedo. Obstaja veliko argumentov, ki bi jih lahko uporabili za izboljšanje našega iskanja (tj. hasCoordinate, country, continent itd.), vendar jih na tej točki ne bomo uporabili.
Če potrebujemo informacije o posamezni funkciji, pred njeno ime natipkamo znak za vprašaj (?) in izpustimo narekovaje. Ko ta ukaz zaženemo, se nam bo odprlo podokno Help z dokumnetacijo za izbrano funkcijo.
Spodnja koda bo najprej odprla dokumentacijo za funkcijo occ_search() in nato shranila njen rezultate v objekt physalus_gbif, ki bi se moral pojaviti v vašem globalnem okolju kmalu po zagonu kode.
Kopirajte in prilepite spodnjo kodo v R skript, premaknite kazalec na prvo vrstico s kodo in kliknite Run. Raziščite dokument, ki se bo odprl na zavihku Help.
Kliknite Run za drugo vrstico kode in preverite delovno okolje: ali je bil ustvarjen nov objekt? Ker bomo vsi vzpostavljali stik s strežnikom GBIF hkrati, lahko traja nekaj časa, da se vse zahteve obdelajo in dokončajo.
?occ_search
physalus_gbif <- occ_search(scientificName = "Balaenoptera physalus")Desno od imena physalus_gbif lahko vidite kakšen tip R objekta je: to je seznam s 5 elementi. Če kliknete majhno modro ikono levo od physalus_gbif, bo njegova vsebina prikazana spodaj. Vidite lahko, da smo dejansko prenesli veliko podatkov. Obstajajo trije seznami, imenovani meta, hierarchy in media (daleč spodaj), in tabela poimenovana data. Peti element sploh ni naveden, saj imajo prvi štirje toliko vsebine, da njegova vsebina ni prikazana. Lahko bi raziskali vse te elemente, vendar bomo pogledali samo del data, kjer so shranjene koordinate in druge informacije o pojavljanjih brazdastega kita.
V tabeli data je 500 opažanj braszdastih kitov, kar pa ni nujno dejansko število pojavljanj v GBIF bazi podatkov. Za preprečitev (pre)obremenitve strežnika je privzeto prenesenih le 500 pojavljanj. Za brazdaste kite je v GBIF bazi podatkov kar 113321 pojavljanj (preverite lahko tukaj). Če želimo spremeniti število prenesenih pojavljanj, dodamo argument limit = in izberemo želeno število (največja omejitev prenosa je 100000 pojavljanj).
Na zaslonu lahko vidite znak za dolar ($) pred imenom elementa. To ni naključje, saj nam pove, da lahko do tega dela seznama dostopamo tako, da za njegovim imenom dodamo $data. Rezultat te operacije bomo shranili v še en nov objekt, ki ga bomo poimenovali physalus_data. To je tabela, v kateri so vsi potrebni podatki za nadaljevanje vaje.
Kopirajte in prilepite spodnjo kodo v R skript, premaknite kazalec na prvo vrstico s kodo in kliknite Run. Preverite svoj delovni imenik, če se po drugem ukazu v njem nahaja novo ime.
Kliknite na ime physalus_data, da se vam odpre tabela v pregled. V naslednjih nekaj minutah raziščite podatke in razmislite, ali so kateri od stolpcev še posebej pomembni za nadaljnje delo.
physalus_gbif <- occ_search(scientificName = "Balaenoptera physalus",
limit = 1500)
physalus_data <- physalus_gbif$data Vprašanja v razmislek:
- Ali je
physalus_datares tabela (ali tibble), kot je bil element$datavphysalus_gbif? - Koliko vrstic in stolpcev ima tabela
physalus_data?
Imate vi kakšno vprašanje?
Izbiranje stolpcev in filtriranje podatkov
Vse v R-u je mogoče narediti na več načinov. Mi si bomo ogledali filtriranje (z uporabo pogoja in ohranitev podmnožice opazovanj – vrstic) in izbiranje (ohranjanje samo ključnih stolpcev) delov naših podatkov s paketom dplyr. R paket dplyr je zelo primeren za R začetnike, saj je zelo intuitiven za uporabo, a kljub temu zelo uporaben in zmogljiv. Spomnite se od začetka vaj, da moramo paket najprej inštalirati in nato aktivirati, da lahko uporabimo njegove funkcije.
Kopirajte in prilepite spodnjo kodo v R skript, premaknite kazalec na prvo vrstico s kodo in kliknite Run. Ponovite še za drugo vrstico.
install.packages("dplyr")
library(dplyr)Za izbiranje stolpcev iz tabel uporabljamo funkcijo select(), ki potrebuje vsaj dva argumenta: ime podatkovnega objekta, iz katerega želimo izbrati, in ime vsaj stolpca, ki ga želimo izbrati. S select() lahko izberemo več stolpcev naenkrat. Od več kot 100 stolpcev, ki smo jih prenesli iz baze GBIF, imajo le nekateri v tem trenutku za nas pomembne podatke: npr. stolpca s koordinatami (decimalLatitude, decimalLongitude), stolpec z državo pojavljanja (country), stolpec z napako v koordinatah (coordinateUncerttaintyInMeters).
Za filtriranje po vrednostih vrstic uporabljamo funkcijo filter(), ki prav tako sprejme dva argumenta: ime podatkovnega objekta, ki ga želimo izbrati iz in izraz, na podlagi katerega želimo filtrirati. Na primer, ne želimo opažanj brazdastih kitov z veliko napako, kajne? Če želimo obdržati samo tiste s 5 km ali manj, moramo preprosto določiti ime stolpca coordinateUncerttaintyInMeters in pogoj filtriranja (< 5000). 5 km ni tako slabo glede na gibalne sposobnosti kitov plavutatih, kajne? Rezultate izbiranja stolpcev in filtriranja bomo shranili v objekt physalus_data, kar bo prepisalo izvirni objekt.
physalus_data <-
select(physalus_data,
species,
decimalLatitude,
decimalLongitude,
coordinateUncertaintyInMeters,
country)
physalus_data <-
filter(physalus_data,
coordinateUncertaintyInMeters < 5000)Funkciji select() in filter() imata zelo generični imeni in sta definirani v različnih R paketih. Odvisno od tega, kateri paket smo nazadnje aktivirali s funkcijo library(), R izbere verzijo funkcije, ki jo bo uporabil. Lahko se zgodi, da po zagonu kode ne bomo dobili željenih rezultatov. V tem primeru je zgornji razlog napogojsteši krivec.
Če se želimo izogniti tem težavam, lahko R-u a priori povemo funkcijo iz katerega paketa želimo uporabiti, tako da pred funckijo napišemo ime paketa in dvojno dvopičje. Na primer, select() ali filter() iz paketa dplyr:
dplyr::filter(),dplyr::select().
Interaktivni zemljevidi s paketom leaflet
Paket leaflet nam omogoča enostavno kreiranje interaktivnih zemljevidov. Osnovna funkcija, s katero zaženemo tak zemljevid je leaflet() in znotraj oklepajev navedemo, kateri podatkovni objekt želimo prikazati na zemljevidu. Sloje zemljevidu dodajamo tako, da na koncu vrstice uporabimo operator %>%, kar R razumemo kot: k prejšnjim ukazom dodaj tudi naslednjega. S funkcijo addProviderTiles() določimo ozadje našega zemljevida. Izbiramo lahko med številnimi možnostmi, ki si jih lahko ogledate tu. Za začetek bomo uporabili ozadje "Esri.WorldImagery", tako da definiramo provider = "Esri.WorldImagery". Lahko pa ga enostavno tudi zamenjamo, npr. s provider = "OpenTopoMap". Nato ponovimo operator %>% in s funkcijo addCircleMarkers() dodamo točke na naš zemljevid. Ta funkcija zahteva, da določimo, kateri stolpcev v podatkih je geografska dolžina (lng = ~) in kateri geografska širina (lat = ~). Leaflet zahteva, da pred imeni stolpcev iz tabele dodamo znak ~.
Kopirajte in prilepite spodnjo kodo v R skript, premaknite kazalec na prvo vrstico s kodo in kliknite Run. Premaknite kazalec na vsako vrstico, ki vsebuje kodo, in ponovite. Koda, ki se začne z leaflet(physalus_data), se mora zagnati od začetka do konca po enem kliku na Run.
install.packages("leaflet")
library(leaflet)
leaflet(physalus_data) %>%
addProviderTiles("Esri.WorldImagery") %>%
addCircleMarkers(
lng = ~ decimalLongitude,
lat = ~ decimalLatitude)Zemljevid pojavljanj lahko še malo vizualno uredimo. Na primer, spremenimo barvo, prosojnost ali velikost točk, ali pa odstranimo obrobo. Vsaka od teh lastnosti je definirana s svojim argumentom, ki ga dodamo znotraj oklepajev funkcije addCircleMarkers():
- s
color =lahko spremenimo barvo točk. Barve, ki jih lahko uporabimo najdete tu, delujejo pa tiste, ki nimajo števila na koncu imena. - z
radius = 5lahko spremenimo velikost točk, uporabimo lahko pozitivna števila, - s
stroke = FALSElahko odstranimo obrobo točk, - s
fillOpacity = 0.7spreminjamo transparentnost oziroma prosojnost točk, izberemo lahko vrednosti med 0 in 1, - z
label =pa lahko dodamo oznako točkam, ki bo prikazana, ko bomo z miškinim kazalcem šli čez posamezno točko.
Kopirajte in prilepite spodnjo kodo v R skript, premaknite kazalec na prvo vrstico s kodo in kliknite Run. Koda, ki se začne z leaflet(physalus_data), se mora zagnati od začetka do konca po enem kliku na Run.
leaflet(physalus_data) %>%
addProviderTiles("Esri.WorldImagery") %>%
addCircleMarkers(
lng = ~ decimalLongitude,
lat = ~ decimalLatitude,
color = "deepskyblue",
stroke = FALSE,
radius = 8,
fillOpacity = 0.7,
label = ~country
)Zemljevid bomo izvozili v html obliko, kar bo ohranilo njegove interaktivne lastnosti:
- Klik na gumb
Export
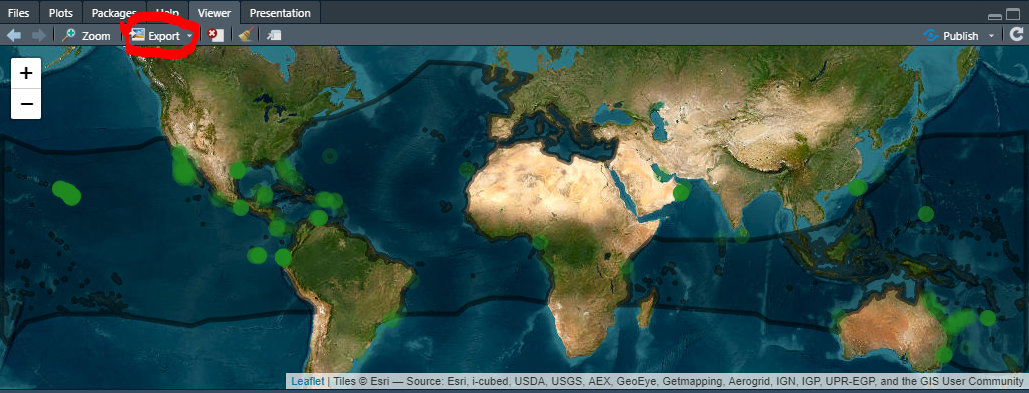
- Izberemo
Save as web page
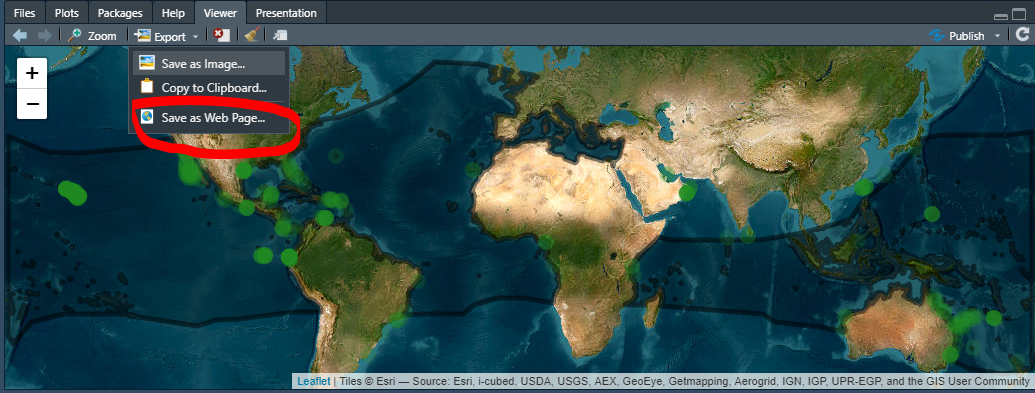
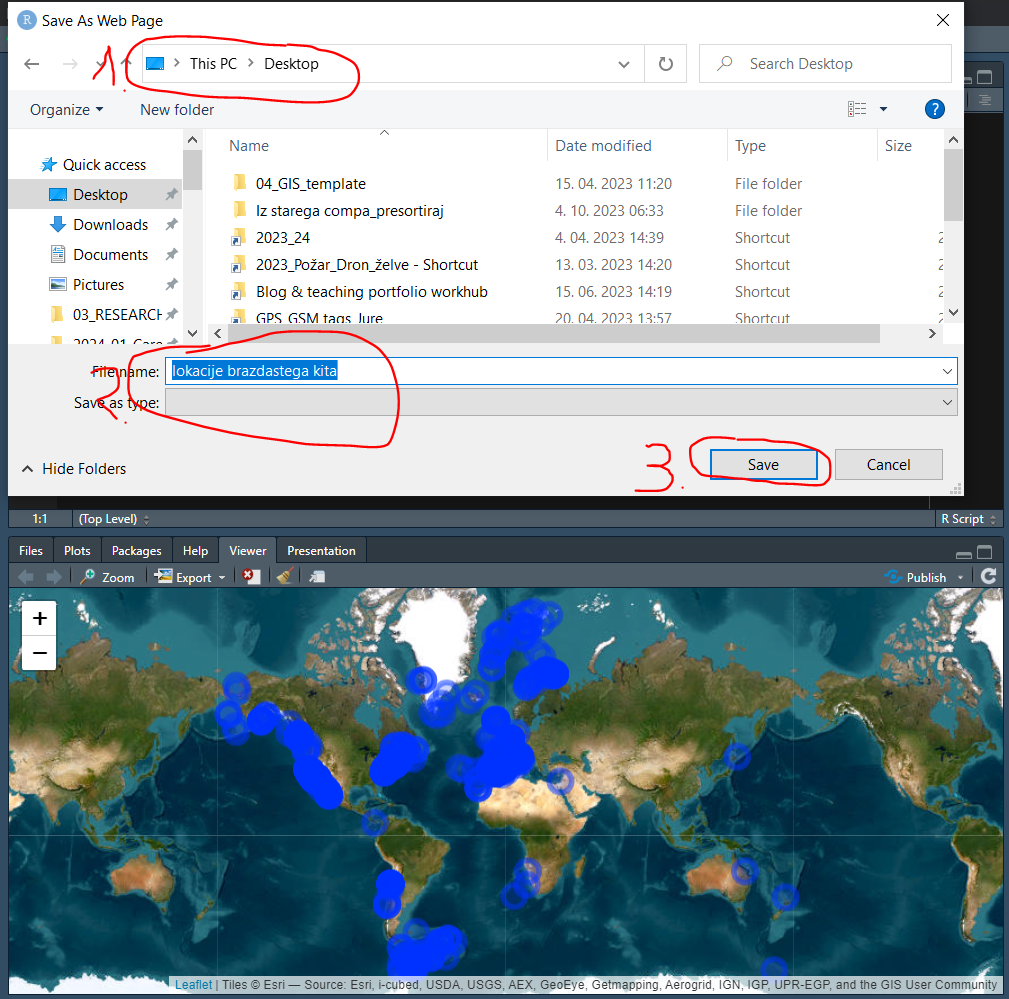
- Določimo ime datoteke in shranimo na namizje našega računalnika
Vprašanja za razmislek:
- Kaj je GBIF in kako je lahko koristen za vaše raziskave/poučevanje?
- Kakšne so pomanjkljivosti podatkov GBIF glede na to, da nekateri od njih izvirajo iz državljanske znanosti (citizen science)?
- Kolikokrat morate zagnati funkcijo
install.packages()in kolikokrat funkcijolibrary()? - Kateri paket in funkcije so enostavni za uporabo za filtriranje in izbiro samo podatkov, ki vas zanimajo?
- Kateri paket in funkcije so enostavni za preprosto vizualizacijo prostorskih točk?
Imate vi kakšno vprašanje?
Če nekaj ne deluje takoj, najprej razmislite o težavi/napaki, se posvetujte s sosedom na levi, nato na desni. Če po tem še vedno ne veste, kako odpraviti težavo ali nadaljevati, vprašajte google ali chat gpt in šele nato vprašajte mene za pomoč!
Današnja naloga bo bolj zahtevna kot zadnjič. Sledite naslednjim korakom, da jo dokončate:
Izberite si eno poljubno vrsto (lahko vašo najljubšo) in poiščite njeno latinsko ime.
Odprite nov R skript v meniju
Filetako, da klikneteNew filein potemR script. Skript bo imel ime Untitled1 (ali Untitled2, če je že odprt prazen skript). Shranite skript na namizje vašega računalnika in ga poimenujteDostop do podatkov iz portala GBIF z R: ime vaše vrste.Kopirajte in prilepite vso kodo, ki smo jo zagnali za primer braszdastega kita v prazno skripto. Vrstice, ki vsebujejo funkcije
install.packages()lahko označite z znakom#, da se ne izvedejo ponovno, saj so paketi zdaj že inštalirani na vašem računalniku.Prilagodite kodo tako, da zamenjate
Balaenoptera physalusz izbranim imenom vrste in preimenujetephysalus_gbifglede na ime vaše vrste (npr.Liza aurata&aurata_gbif).Ponastavite argument
limit =v funkcijiocc_search()in prenesite več kot 500 pojavljanj vrste (500 oz 1500 spremenite v številko po vaši izbiri).Iz podatkovna seznama, ki se konča s končnico
_gbifdostopajte do elementadatain ga shranite v nov objekt. Preimenujte glede na svojo vrsto (npr.aurata_data).- če je število pojavljanj nič ali zelo majhno (< 100), ponovite postopek od 1. koraka.
- če je > 100 pojavitev vaše vrste, nadaljujte s 7. korakom.
Izberite samo smiselne stolpce iz podatkovnega okvira
*_data.Filtrirajte svoje podatke in poskusite spremeniti vrednost filtriranja napake koordinat, da bo bolje ustrezala vaši vrsti (npr. 500 metrov je morda bolje za Liza aurata).
Prikažite filtrirana pojavljanja vaše vrste na
leafletzemljevidu. Raziščite razpoložljive osnovne zemljevide tukaj in ga spremenite v enega po svoji izbiri (npr. zamenjajte"Esri.WorldImageryz drugim imenom iz seznama na spletni strani).Spremenite izgled točk na vašem zemljevidu, tako da poljubno spremenite vrednosti argumentov
color =,radius =infillOpacity =.Izvozite zemljevid v html dokument in ga naložite v nalogo na e-učilnico (lahko danes, lahko do postavljenega roka: 15.11.2023).
Shranite obe R skripti in si ju naložite na USB ali pošljite preko e-maila, da ju lahko ponovno uporabite.
- Na
leafletzemljevidu prikažite mesta iz prvih vaj in spremenite izgled točk, oznaka točke naj bo ime mesta:
ime_mesta <- c("Ljubljana", "Maribor", "Koper", "Kranjska Gora", "Novo mesto")
geografska_sirina <- c(46.047766, 46.550613, 45.547377, 46.4887160, 45.802626)
geografska_dolzina <- c(14.507420, 15.645263, 13.730270, 13.787491, 15.164192)
prebivalstvo <- c(279631, 94370, 25753, 1452, 24183)
primorska <- c(FALSE, FALSE, TRUE, FALSE, FALSE)
mesta <- data.frame(ime_mesta, geografska_dolzina, geografska_sirina, prebivalstvo, primorska)- Na
leafletzemljevidu prikažite domače kraje sošolcev iz prvih vaj in spremenite izgled točk, oznaka točke naj bo ime sošolca:
imena_sosolcev <- c("___", "___", "___", "___")
starost <- c(___, ___, ___, ___)
barva_oci <- c(___, ___, ___, ___)
domaci_kraj <- c("___", "___", "___", "___")
geografska_sirina_dk <- c(___, ___, ___, ___)
geografska_dolzina_dk <- c(___, ___, ___, ___)
sosolci <- data.frame(___, # imena
___, # starosti
___, # barva oci
___, # domaci kraj
___, # geografska sirina
___) # geografska dolzina
sosolci